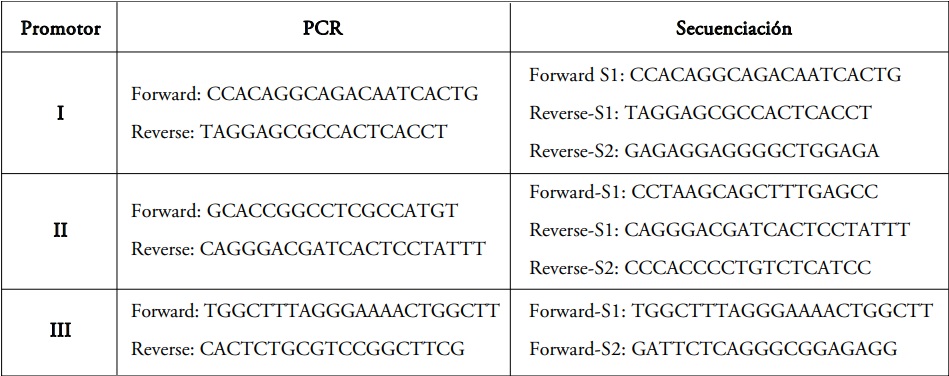
Tabla 1.Cebadores utilizados en reacciones de amplificación y secuenciación de los promotores I, II y III del gen CAST bovino
ARTÍCULO DE INVESTIGACIÓN
Análisis comparativo de promotores del gen de calpastatina en Angus y Brahman
Motter, MM1; Corva, PM2; Soria, LA1.
1
Universidad de Buenos Aires. Facultad de Ciencias Veterinarias, Cátedra de Genética.
2
Universidad Nacional de Mar del Plata,
Facultad de Ciencias Agrarias, Unidad Integrada Balcarce, Departamento de Producción Animal.
Recibido:27/08/2021
Aceptado: 01/12/2021
Correspondencia e-mail:Liliana A. Soria lsoria@fvet.uba.ar
Resumen
El gen de la calpastatina bovina (CAST) se expresa en músculo esquelético generando diferentes isoformas de ARNm a partir de tres promotores (I, II y III), splicing y poliadenilación alternativos. Su expresión varía entre razas y músculos, lo que es relevante porque contribuye a la variabilidad de la terneza de la carne. El objetivo de este trabajo fue analizar las secuencias de los tres promotores de CAST en toros Angus (n=7) y Brahman (n=7) con el propósito de identificar polimorfismos de ADN, potenciales sitios de unión a factores de transcripción (TFBS o transcription factor binding site) e islas CpG. Se identificaron trece SNP, de los cuales cuatro alterarían potenciales TFBS en el promotor I (sitios NF-кB en las posiciones 96.034.491, 96.034.660 y 96.034.661 de BTA7 del genoma bovino de referencia ARS UCD 1.2 y sitios E2F o Sp1 en la posición 96.034.610) y tres en el promotor II (96.034.941 sitio Sp1 y 96.035.456 y 96.035.504 sitios E2F). Se identificaron islas CpG en los tres promotores y SNPs en I y II que alterarían dinucleótidos CpG. Estos resultados plantean nuevas hipótesis y objetivos para la investigación en la calidad de la carne, y la necesidad de estudios complementarios para validarlos.
Palabras clave:bovinos para carne, calpastatina, factores de transcripción, islas CpG
Comparative analysis of promoters of the calpastatin gene in Angus and Brahman
Summary
The bovine calpastatin (CAST) gene has different isoforms expressed in skeletal muscle that originate from three promoters (I, II and III), with alternative splicing and variable polyadenylation sites. Total and relative expression of isoforms vary between breeds and muscles, contributing to the variability of the meat tenderness. The objective of this study was to analyze the sequence of the three CAST promoters in Angus (n = 7) and Brahman (n = 7) bulls in order to identify DNA polymorphisms, potential transcription factor binding sites (TFBS) and CpG islands. Thirteen SNPs were identified, four of them would modify potential TFBS in promoter I (NF-кB site: positions 96,034,491, 96,034,660 and 96,034,661 and E2F or Sp1 sites I position 96,034,610 on BTA7; reference genome ARS UCD 1.2) and three in promoter II (in 96,034,941 Sp1 and 96,035,456 and 96,035,504 E2F sites). CpG islands were identified in the three promoters and SNPs in I and II that would alter CpG dinucleotides. These results could lead to new hypotheses and objectives in the investigation of meat quality, particularly about differences among cattle breeds.
Key words: beef cattle, calpastatin, transcription factor, CpG islands
INTRODUCCIÓN
La calpastatina es el inhibidor endógeno específico del sistema de las calpaínas, el
cual participa en la degradación de las proteínas miofibrilares tanto en el tejido muscular
in vivo como postmortem, y además regula el
recambio de proteínas y la diferenciación celular en el tejido muscular y por lo tanto el
crecimiento en general12. El gen de la calpastatina bovina (CAST) tiene una regulación de la
expresión altamente compleja porque depende de cuatro promotores; tres de los cuales
(I, II y III) son activos en músculo esquelético
dando origen a mensajeros con extremo 5’ en
los exones 1xa, 1xb y 1u, respectivamente23,
dicho gen presenta además, splicing y poliadenilación alternativos2,23,24.
Las razas índicas y sus cruzas se utilizan
en sistemas de producción extensiva tropical y
subtropical porque tienen mayor adaptabilidad,
tolerancia al calor y a garrapatas, longevidad y fertilidad que las razas europeas; sin embargo producen carne de menor calidad que estas últimas.
Está confirmado que la raza Brahman tiene mayor
actividad de calpastatina en el músculo en comparación con Angus, considerándose como uno de
los factores que determina la menor terneza de la
carne de dicha raza22.
Se han realizado estudios de la expresión
del gen CAST en distintas razas y músculos, hallándose diferencias significativas en el nivel de
expresión de las distintas isoformas de mensajeros de CAST15,16,17, pero aún no hay consenso sobre
la relación específica entre la forma de expresión
de dicho gen y la magnitud de su efecto sobre la
terneza según músculo o raza.
Los promotores alternativos de un gen
son responsables de la expresión génica específica de tejido o de una etapa de desarrollo. Algunos posibles mecanismos que explican cómo los
múltiples promotores son utilizados selectivamente incluyen: distinta estructura del core del
promotor, sitios de inicio de transcripción dispersos regulados por proteínas de unión específicas al ADN, como factores Sp1 o NF-Y9, presencia de elementos cis-reguladores distales tales
como enhancers13 o presencia de componentes
tejido-específico homólogos a factores de transcripción basal5. Otro mecanismo relevante de
control de la expresión génica es el epigenético, el cual incluye, entre otros, a la metilación
del ADN, el cual es un mecanismo de silenciamiento génico que consiste en la adición de un
grupo metilo al C5 de una citosina (5-mC) que
precede a una guanina (CpG). Con frecuencia, estos dinucleótidos se encuentran agrupados
en regiones denominadas islas CpG. La metilación se ha asociado con una disminución de la
actividad transcripcional ya que puede inhibir
la unión de los TFIIs (transcription factor RNA
polymerase II) de manera directa o indirecta, reclutando proteínas de unión a metil-CpG (MBP,
methyl binding protein), que poseen actividades
represivas de remodelación de la cromatina10.
Los patrones de metilación pueden ser
inducidos por diversos factores, tales como edad,
manejo, infecciones bacterianas, entre otros, lo
cual indicaría que en algunos casos, la metilación
del ADN es un proceso dinámico de respuesta a
estímulos externos8.
La expresión génica también puede resultar afectada por la presencia de polimorfismos (SNPs o Indels) en los promotores, tanto
por alterar TFBS como las islas CpG y su potencial metilación18, 29.
Dada la relevancia del control de la transcripción sobre el nivel y el perfil (isoformas) de la
expresión del gen CAST, el objetivo del presente
trabajo fue analizar y comparar las secuencias de
tres promotores (I, II y III) del gen CAST en las razas Angus y Brahman en busca de polimorfismos
que pudieran alterar sitios de unión a factores de
transcripción y/o islas CpG.
MATERIALES Y MÉTODOS
Muestras
A partir de muestras de semen o sangre (extraída con EDTA como anticoagulante) de 14 toros (7 Angus y 7 Brahman) de uso en inseminación artificial en Argentina, se aisló el ADN mediante un método estándar de fenol/cloroformo25, agregándose 0.4M ditiotreitol (DDT) a la solución de lisis celular en las muestras de semen.
Análisis Molecular
La secuencia de nucleótidos de los promotores I, II y III de CAST se recuperaron de la base de datos GenBank (Nro. de acceso AH014526). Se diseñaron cebadores específicos mediante el programa Primer3 que fueron utilizados en las reacciones de PCR y de secuenciación (Tabla 1). Se amplificaron fragmentos que contienen las regiones potenciales de dichos promotores, descriptas por Raynaud et al23. Para cada reacción de PCR a 100 ng de ADN se le agregaron 0,25 μM de cada cebador, 200 μM de cada dNTP (Promega Corporation, WI, USA), 1,5 mM de Cl2 Mg, 2U de Kapa2G Fast DNA Polymerase (Sigma-Aldrich, St. Louis, USA), 5μl de Buffer 1X y H2 O en cantidad suficiente para 25 μl de volumen final. Cada reacción se realizó en un termociclador marca Mastercycler personal (Eppendorf, Stevenage, UK), en 35 ciclos y con el siguiente programa: 20 seg a 94 °C, 15 seg a la temperatura de annealing específica para cada par de cebadores (58 °C, 56 °C y 59 °C para I, II y III, respectivamente), 15 seg a 72 °C, con un primer paso a 95 °C durante 5 min y una extensión final durante 5 min a 72 °C. Los amplicones obtenidos se visualizaron en geles de agarosa al 2 % teñidos con GelRed® Nucleid Acid Gel Stain (Biotium, Fremont, USA) y posteriormente se purificaron con PureLink® Quick Gel Extraction and PCR Purification kit (Invitrogen Life Technologies, Brasil) según protocolo del fabricante. Los amplicones purificados fueron secuenciados en un ABI3130XL Genetic Analyzer (Applied Biosystems, Foster City, USA) en el Servicio de Secuenciación de la Facultad de Ciencias Exactas y Naturales de la Universidad de Buenos Aires, con los cebadores diseñados para tal fin (Tabla 1).
Análisis Bioinformático
Las secuencias de nucleótidos obtenidas
fueron editadas mediante el programa BioEdit Sequence Alignment Editor y se alinearon con el programa ClustalW Multiple Alignment.
Para identificar potenciales TFBS, se
utilizó el programa Matinspector, versión 11.3
(Genomatix GmbH, München, Alemania) con
las siguientes restricciones: Grupo de matrices: elementos promotores centrales generales
y vertebrados; Similitud de core: 0.9, Similitud
de matrices: optimizado. Dicho programa utiliza matrices de peso posicional (PWM, position
weight matrix) construidas a partir de sitios de
unión de factores de transcripción experimentalmente determinados.
El análisis de la presencia de islas CpG en
las tres regiones promotoras del gen CAST se realizó mediante el programa Methyl Primer Express,
version 1.0 (Applied Biosystems, Foster City, USA).

Tabla 1.Cebadores utilizados en reacciones de amplificación y secuenciación de los promotores I, II y III del gen CAST bovino
RESULTADOS
Los SNP hallados en las secuencias alineadas de los tres promotores de CAST de los 14
toros analizados se resumen en la Tabla 2. Dicha
comparación permitió identificar haplotipos diferentes en los promotores I y II en cada raza analizada, no hallándose diferencias en el promotor III.
En la muestra de toros Angus se halló el haplotipo
CTCCA en el promotor I y el haplotipo GCTGG en
las secuencias aisladas de los toros Brahman. Al
promotor II le correspondieron el haplotipo CCG
en Angus y el GTC en Brahman (Tabla 2).
El análisis de predicción de los potenciales elementos regulatorios en cis o TFBS permitió identificar los factores que interactúan
potencialmente con las tres regiones promotoras y establecer que algunos de los ellos podrían
ser alterados por los SNP hallados en la comparación de secuencias de Angus y Brahman (Tabla 2 y Fig. 1, 2 y 3).
De los resultados obtenidos del análisis
de predicción de TFBS pudo observarse que en el
promotor I, los SNP de las posiciones 96.034.491,
96.034.660 y 96.0346.61 de BTA7 (posiciones
-70, +101, +102 respectivamente con respecto
al sitio de inicio de la transcripción) crearían
sitios de unión al factor NF-кB (Nuclear Factor
kappa Beta) cuando se presenta el alelo hallado
en las secuencias obtenidas de Brahman (G en
las tres posiciones) (Fig. 1). El SNP de la posición 96.034.610 (+51) modificaría un TFBS que
potencialmente podría unirse a dicha secuencia;
en el caso del alelo C (hallado en Angus) sería un
sitio E2F, mientras que con el alelo T (hallado en
Brahman) sería un sitio Sp1.
En la secuencia correspondiente al promotor II se predijo un sitio Sp1 en la región que
contiene al SNP 96.034.941 (-637) en presencia
del alelo G (hallado en Brahman). También en la
secuencia de dicho promotor, los SNP de las posiciones 96.035.456 (-122) y 96.035.504 (-74)
se predijeron sitios de unión a E2F cuando están
presentes los alelos hallados en Angus (T y C, respectivamente) (Fig. 2A y B). Se pudo determinar
también una región rica en sitios Sp1 y E2F alrededor del SNP 96.035.456, particularmente entre
las posiciones -139 a -91, donde se identificaron
seis sitios Sp1 en ambas razas, dos E2F en Brahman y tres en Angus (Fig 2B).
El análisis realizado para identificar islas
CpG en los tres promotores, determinó que las
mismas podrían abarcar amplias regiones distales
de los promotores I y III (559 pb y 619 pb, respectivamente) y la región completa analizada en
el promotor II (Fig. 1, 2 y 3). Alguno de los SNP
identificados en los promotores I y II podrían alterar algún dinucleotido CpG, y por lo tanto también
el patrón potencial de metilación de la isla CpG determinada (Fig. 1 y 2).
Tabla 2. SNP hallados en los promotores I, II y III del gen CAST en Angus y Brahman y los potenciales TFBS que modifican
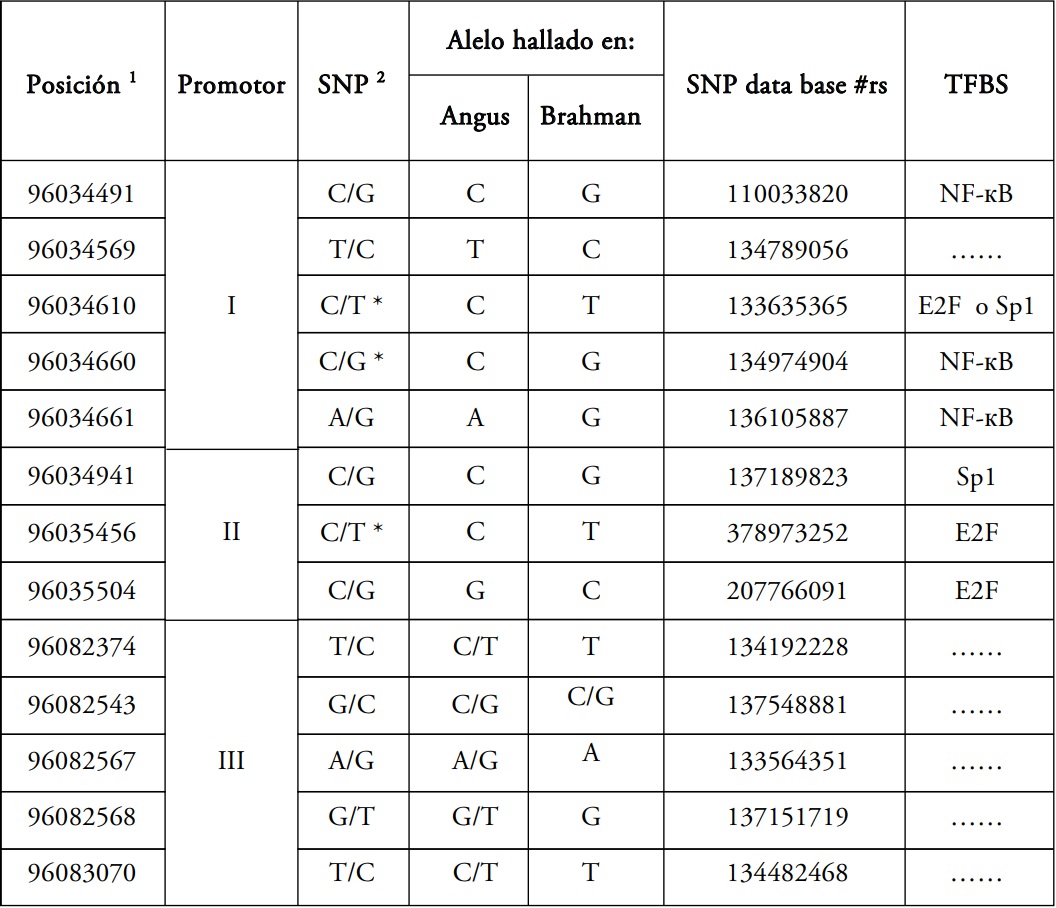
1 Posición sobre el cromosoma 7 (BTA7) del genoma bovino de referencia ARS UCD 1.2.
2 Los polimorfismos que podrían modificar un sitio CpG se indican con asterisco.

Figura 1.Potenciales TFBS en el promotor I del gen CAST predichos por análisis bioinformático y SNP identificados por
secuenciación en Angus y Brahman.
Ref.: Las posiciones con respecto al sitio de inicio de la transcripción de los SNP detallados en la Tabla 2 (en la figura, señalados con recuadros) son respectivamente: -70: S=C/G; +8: y=T/C; +51: y=C/T y +101/+102: sr=CA/GG. Los TFBS que
pueden variar entre razas se indican con línea punteada. Con línea entera se indica la isla CpG. El extremo 5’ del exón 1xa se
indica con letras minúsculas.
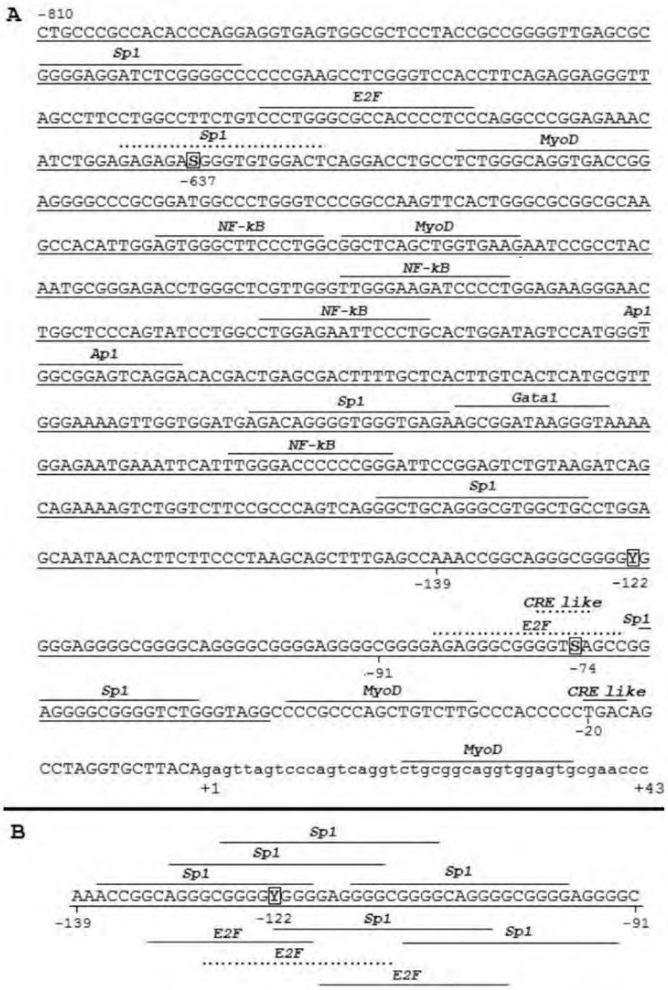
Figura 2.Potenciales TFBS en el promotor II del gen CAST predichos por análisis bioinformático y SNP identificados por secuenciación
en Angus y Brahman (A) y en la secuencia delimitada entre los nucleótidos -139 y -91 (B).
Ref.: Las posiciones con respecto al sitio de inicio de la transcripción de los SNP detallados en la Tabla 2 (en la figura, señalados con recuadros) son respectivamente: -637 S=C/G, -122 Y=C/T y -74 S=G/C. Los TFBS que pueden variar entre razas se indican con línea punteada.
Con línea entera se indica la isla CpG. El extremo 5’ del exón 1xb se indica con letras minúsculas.

Figura 3.Potenciales TFBS en el promotor III del gen CAST predichos por análisis bioinformático y SNP identificados por secuenciación en Angus y Brahman.
Ref.: Las posiciones con respecto al sitio de inicio de la transcripción de los SNP detallados en la Tabla 2 (en la figura, señalados con recuadros) son respectivamente: -750 Y=T/C, -581 S=G/C, -561/-562 RK=AG/GT y -145 Y=T/C. Con línea entera
se indica la isla CpG. El extremo 5’ del exón 1u se indica con letras minúsculas.
DISCUSIÓN
La expresión de CAST se regula a partir de la combinación integrada de la actividad
del promotor, el splicing y la poliadenilación
alternativa2,23,24, generando de ese modo, distintas isoformas de mensajeros. Lejos de ser una
excepción, la regulación de la expresión génica
mediante la generación de numerosas isoformas es un proceso usual, acoplado y regulado
en forma conjunta11,14.
Las regiones promotoras del gen CAST
son ricas en islas CpG, carecen de caja TATA y secuencias regulatorias proximales del tipo CAAT,
características propias de genes housekeeping, los
cuales suelen presentar múltiples sitios Sp123.
Los TFBS más abundantes y comunes
identificados en los tres promotores son Sp1 y
NF-кB. Sp1 es un factor de transcripción del tipo
“dedos de zinc” (Zinc-fingers) que se une a motivos ricos en GC de muchos promotores, y es
un factor de transcripción que puede activar la
transcripción en ausencia de caja TATA23. Sp1 y
NF-кB participan en muchos procesos celulares,
incluida la diferenciación y crecimiento celular,
respuestas inmunitaria e inflamatoria y la remodelación de la cromatina21.
Los SNP hallados en el promotor I en las
secuencias aisladas en Brahman, revelaron que
podrían crear dos sitios NF-кB y un sitio Sp1. Estos
SNP están ubicados en la región más importante
para la activación de la expresión de la isoforma I
de CAST23, pudiendo ser una de las posibles causas
en las diferencias de actividad de calpastatina halladas entre razas bovinas22.
La secuencia del promotor II posee una
alta concentración de guaninas lo que resulta
en múltiples sitios Sp1. El alelo G del SNP presente en Brahman en la posición -637 crearía
un sitio Sp1. Por otra parte, los SNP -122 y -74
modifican sitios de unión para E2F (Fig. 2), el
cual entre otras funciones, es un regulador del
metabolismo en condiciones no proliferativas6.
Estudios de regulación realizados sobre muestras de músculo cardíaco bovino mostraron un
aumento en la transcripción de calpastatina en
respuesta a ß-agonistas por la vía de señalización dependiente de la proteína kinasa A (PKA),
indicando dos posibles sitios de respuesta a
AMPc en la región -76 (GGTCA) y -20 (TGAC)
del promotor II, los cuales han sido denominados sitio “CRE (cAMP response element) like” o
“CRE like half” (porque estos sitios no presentan la secuencia palidrómica conservada típica
de los sitios CRE, que es: TGACGTCA)2 . El hallazgo de una C en el SNP de la posición -74 del
promotor II, en toros Brahman, sugiere que en
dicha región se podría originar un TFBS del tipo
“CRE like”. Los ß-agonistas son utilizados como
promotores del crecimiento y la eficiencia alimentaria, pero también afectan la calidad de la
carne, principalmente reduciendo la terneza4,
efecto que está relacionado con el aumento de
la actividad de calpastatina27.
En la comparación de expresión de
CAST entre Angus y Brahman, Nattrass et al17
hallaron interacciones significativas entre raza
y administración de ß-agonistas, y entre raza y
sexo, lo que señala que los promotores del crecimiento y las hormonas sexuales tienen efecto
sobre la expresión de dicho gen y que ese efecto podría estar explicado por cambios en el nivel de la transcripción de CAST dependiente de
TFBS o de mecanismos epigenéticos.
En el promotor II también se identificaron numerosos sitios MyoD, un factor clave en la
diferenciación del tejido muscular esquelético
y el mantenimiento de la capacidad metabólica
oxidativa26, que también puede interactuar con
proteínas involucradas en la remodelación de
la cromatina promoviendo la acetilación de histonas, y en consecuencia la activación génica.
Estudios de expresión realizados en el músculo Longissimus dorsi de bovinos de raza Nelore,
demostraron una menor expresión de MyoD en
muestras de carne más tierna28. La presencia de
estos sitios en el promotor hace posible su unión
y por lo tanto la activación podría depender de la
concentración del factor MyoD.
En cada uno de los promotores analizados
se hallaron sitios GATA1, un factor de transcripción que contiene dos dedos de zinc altamente
conservados, y aunque solo tres subtipos de factores de transcripción GATA (GATA 4, 5 y 6) se
expresarían en tejido de origen mesodérmico y
endodérmico, se ha reportado que GATA1 podría
estar regulando la transcripción de CAST23.
Además de la diversidad de promotores,
el control de la expresión génica de CAST incluye
también el splicing y la poliadenilacion alternativa, mecanismos que podrían estar regulados conjuntamente con la transcripción. Se ha demos-trado que el reclutamiento específico de ciertos
factores de la transcripción, la arquitectura del
promotor3
, así como también la fosforilación de
la ARN polimerasa7
influyen en los procesos de
splicing. La poliadenilación alternativa influye en
la vida media de los ARNm, porque aquellos que
poseen 3´UTR más cortos son generalmente más
estables debido a que en esa región presentan
menos sitios de unión a proteínas o microARN
que promueven su degradación, dicho proceso
de poliadenilacion alternativa también podría
estar regulado, entre otros factores, por secuencias presentes en el promotor19.
Se han hallado diferencias en la expresión de CAST entre razas bovinas18, entre distintos
músculos bovinos15,16,17, como así también se han
identificado mensajeros alternativamente poliadenilados en distintos músculos bovinos1,17.
Motter el al15, hallaron que las isoformas
de CAST que más se expresan en tres músculos
(infraspinatus, triceps brachii y semitendinosus)
de novillos Angus son la II y la III, y además que
la isoforma III tiene una variante que excluye al
exón 3, siendo la isoforma III con exón 3 la más
abundante en el músculo semitendinosus. En
cambio, al realizar el mismo análisis en músculos de novillos Brahman, se halló un resultado
inverso con relación a la expresión de la isoforma III en semitendinosus, donde la isoforma más
abundante fue la isoforma III sin exón 3 (resultados inéditos).
Con relación a mensajeros de CAST alternativamente poliadenilados, Casale et al1
han
descripto tres tipos de ARNm: cortos, medianos y
largos, al analizar tres músculos bovinos (infraspinatus, triceps brachii y semitendinosus) de dos
razas (Angus y Brahman).
Todos estos resultados sugieren que la
existencia de polimorfismos afectando TFBS
en los promotores de CAST no serían la única
causa probable de diferencias de expresión
entre razas. Deben considerarse también los
cambios a nivel del control epigenético de la
expresión, como podrían ser los cambios en
los patrones de metilación de las extensas islas
CpG identificadas en cada uno de los promotores analizados, las cuales podrían ser blanco de
metilación y por lo tanto, disminuir la expresión de dicho gen. Parr et al20 han demostrado
que en los promotores del gen CAST porcino
existen polimorfismos que modificarían el patrón de metilación de los promotores I y II, lo
cual daría origen a diferencias entre razas en la
expresión de dicho gen.
En síntesis, este trabajo aporta información sobre las potenciales causas que podrían
explicar diferencias en la expresión del gen de
la calpastatina entre dos razas bovinas que han
sido siempre un ejemplo paradigmático de las
diferencias en calidad de la carne. Estudios funcionales (Mobility shift assay, estudios de expresión in vitro, por ejemplo) permitirían confirmar
la interacción los TFBS descriptos, la existencia
de diferencias en los patrones de metilación de
las islas CpG delimitadas y la potencial influencia
de los SNP identificados sobre la actividad de los
tres promotores de CAST.
CONCLUSIÓN
El análisis de las secuencias de los tres promotores de CAST activos en músculo esquelético en toros Angus y Brahman permitió identificar trece polimorfismos del tipo SNP y también confirmar la existencia de una extensa isla CpG en cada uno de ellos. Los SNP identificados podrían alterar potenciales TFBS identificados en cada promotor o incluso alterar el patrón de metilación en las islas CpG de los promotores I y II. Estos hallazgos contribuyen a la definición de nuevas hipótesis acerca del control de la expresión de un gen sumamente complejo, y que en el caso particular de los animales domésticos tiene un rol clave en la definición de la calidad y valor de la carne.
AGRADECIMIENTOS
Este trabajo fue realizado con subsidios de la Universidad de Buenos Aires (UBACyT 2010-1012-20020090100072 y 2020-2023- 20020190100272). El presente trabajo es parte de la tesis de Mariana M. Motter en cumplimiento parcial de los requisitos para el título de Doctor (Facultad de Ciencias Veterinarias, Universidad de Buenos Aires).
1. Casale, M. F.; Silvestro, C.; Corva, P.M.; Soria, L.A. Alternatively polyadenylated calpastatin transcripts in
bovine muscles. Journal of Basic and Applied Genetics.
2020; 31, (1): 45–51.
2. Cong, M.; Thompson, V. F.; Goll, D. E.; Antin, P. B. The bovine
calpastatin gene promoter and a new N-terminal region of
the protein are targets for cAMP-dependent proteinkinase
activity. Journal of Biological Chemistry.1998; 273: 660-666.
3. Cramer, P.; Cáceres, J.F.; Cazalla, D.; et al. Coupling of transcription with alternative splicing RNA pol II promoters
modulate SF2/ASF and 9G8 effects on an exonic splicing
enhancer. Mol Cell. 1999; 4 (2): 251-258.
4. Cruz, F.L.; de Carvalho, E.B.; Ramos, E.M.; Pereira,
L.J.; Zangeronimo, M.G. Relationship between beta-adrenergic agonists, calpain system activity and
beef texture: A systematic review. J Anim Physiol Anim
Nutr. 2021; 105: 442– 451.
5. Davuluri, R.V.; Suzuki, Y.; Sugano, S.; Plass, C.;
Huang,T.H. The functional consequences of alternative
promoter use in mammalian genomes. Trends Genet.
2008 ;24 (4) :167-177.
6. Denechaud, P.D.; Fajas, L.; Giralt, A. E2F1, a Novel Regulator
of Metabolism. Frontiers in Endocrinology. 2017; 8. 311.
7. Hirose, Y.; Tacke, R.; Manley, J.L. Phosphorylated RNA
polymerase II stimulates pre-mRNA splicing. Genes Dev.
1999; 13 (10):1234-1239.
8. Ju, Z.; Jiang, Q.; Wang, J.; et al. Genome-wide methylation
and transcriptome of blood neutrophils reveal the roles
of DNA methylation in affecting transcription of protein-coding genes and miRNAs in E. coli-infected mastitis
cows. BMC Genomics 2020; 21, 102.
9. Kadonaga, J.T. Perspectives on the RNA polymerase II
core promoter. WIREs Dev Biol, 2012; 1: 40-51.
10. Kaludov, N.; Wolffe, A. P. MeCP2 driven transcriptional repression in vitro: selectivity for methylated DNA, action
at a distance and contacts with the basal transcription
machinery.Nucleic Acids Res. 2000; 1921–1928.
11. Kolathur, K.K. Role of promoters in regulating alternative
splicing, Gene, 2021; 782.
12. . Koohmaraie, M.; Kent, M.P.; Shackelford, S.D.; Veiseth,
E.; Wheeler, T.L. Meat tenderness and muscle growth:
is there any relationship? Meat Science. 2002; 62 (3):
345-352.
13. Levine, M.; Tjian, R. Transcription regulation and animal
diversity. Nature. 2003; 424: 147-151.
14. Mapendano, C. K.; Lykke-Andersen,S.; Kjems, J.;Bertrand,
E.; Jensen, T.H. Crosstalk between mRNA 3′ end processing
and transcription initiation. Mol. Cell 2010; 40:410–422.
15. Motter, M.M.; Corva, P.M.; Soria, L.A. Expression of calpastatin isoforms in three skeletal muscles of Angus
steers and their association with fiber type composition and proteolytic potential. Meat Science. 2021; 171.
16. Muroya S.; Neath, K.E.; Nakajima, I.; et al. Differences
in mRNA expression of calpains, calpastatin isoforms
and calpain/ calpastatin ratios among bovine skeletal
muscles. Animal Science Journal. 2012; 83 (3):252-259.
17. Nattrass, G.S.; Café, L.M.; McIntyre, B.L.; et al. A post-transcriptional mechanism regulates calpastatin expression
in bovine skeletal muscle. Journal of Animal Science.
2014; 92 (2):443-455.
18. Ordovás, L.; Roy, R.; Pampín, S.; et al. The g.763G>C SNP
of the bovine FASN gene affects its promoter activity
via Sp-mediated regulation: implications for the bovine
lactating mammary gland. Physiol Genomics. 2008; 15;
34(2):144-148.
19. Oktaba,K.; Zhang,W.; Lotz, T.S.; et al. ELAV Links paused
Pol II to alternative polyadenylation in the Drosophila
Nervous System. Molecular Cell. 2015; 57 (2):341-348.
20. Parr, T.; Jewell, K.K.; Sensky, P.L.; Brameld, J.M.; Bardsley, R.G.; Buttery, P.J. Expression of calpastatin isoforms
in muscle and functionality of multiple calpastatin promoters. Arch Biochem Biophys. 2004; 427(1):8-15.
21. Pires B.R.B.; Binato, R.; Ferreira, G.M.; Cecchini, R.; Panis, C.; Abdelhay, E. NF-kappaB Regulates Redox Status in Breast Cancer Subtypes. Genes (Basel). 2018; 9
(7):320.
22. Pringle, T. D.; Williams, S.E.; Lamb, B.S.; Johnson, D.D.;
West, R.L. Carcass characteristics, the calpain proteinase system, and aged tenderness of Angus and Brahman
crossbred steers. J. Anim. Sci. 1997; 75:2955–2961.
23. Raynaud, P.; Jayat-Vignoles, C.; Laforet, M.P.; and Leveziel,
H.Four promoters direct expression of the calpastatin gene.
Archives of Biochemistry and Biophysics. 2005a; 437, 69-77.
24. Raynaud, P.; Gillard, M.; Parr, T.; Bardsley, R.; Amarger,
V.; Levéziel, H. Correlation between bovine calpastatin
mRNA transcripts and protein isoforms. Archives of Biochemistry and Biophysis. 2005b; 440: 46-53.
25. Sambrook, J.; Fritsch, E.F and Maniatis, T. Molecular cloning: a laboratory manual, 2nd ed 1989; Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y.
26. Shintaku, J.; Peterson, J. M.; Talbert, E. .; et al. MyoD
Regulates Skeletal Muscle Oxidative Metabolism Cooperatively with Alternative NF-κB. 20. Cell reports, 2016;
17(2):514–526.
27. Strydom, P. E.; Hope-Jones, M.; Frylinck, L.; Webb, E. C. The
effects of a beta-agonist treatment, vitamin D3 supplementation and electrical stimulation on meat quality of
feedlot steers. Meat Science, 2011; 89: 462–468.
28. Tizioto, P.C.; Coutinho, L.L.; Mourão, G.B, et al. Variation
in myogenic differentiation 1 mRNA abundance is associated with beef tenderness in Nelore cattle. Anim Genet.
2016; 47 (4):491-494.
29. Vohra, M.; Adhikari, P.; Souza, S.C.; Nagri, S.K.; Umakanth,
S.; Satyamoorthy, K.; Rai, P.S. CpG-SNP site methylation
regulates allele-specific expression of MTHFD1 gene in
type 2 diabetes. Lab Invest. 2020; 100(8):1090-1101